Informationsblatt 7: p53

 Diese
Seite als PDF herunterladen
[PDF] [198 KB]
Diese
Seite als PDF herunterladen
[PDF] [198 KB]
Informationsblatt 7: p53 - Schutzengel des Genoms
Das Tumorsuppressor-Protein p53 ist ein Transkriptionsfaktor, dessen Gen beim Menschen auf dem kurzen Arm von Chromosom 17 lokalisiert ist. In der Kurzbezeichnung steht der Buchstabe p für Protein und 53 für die Molekülmasse in Kilodalton, wie sie in der Gelelektrophorese ermittelt wurde. Das menschliche p53 ist 393 Aminosäuren lang und besitzt 7 Domänen:
![]()
|
Domäne
|
Position
|
Funktion
|
|
AD 1
|
1-42
|
Aktivierung weiterer
Transkriptionsfaktoren
(z.B. für Gene von DNA-Reparaturenzymen)
|
|
AD 2
|
43-63
|
Aktivierung
der Apoptose
|
|
PR
|
80-94
|
|
|
DBD
|
100-300
|
DNA-Bindungsregion
|
|
OD
|
307-355
|
Bindungsregion für
weitere p53; p53 ist erst nach
Zusammenlagerung von 4 p53-Proteinen funktionstüchtig (= Tetramerisierung)
|
|
NLS
|
316-325
|
Signal für den
Transport in den Zellkern
|
|
DRDB
|
356-393
|
regelt DNA-Bindung nach unten |
Bereits beim Kapitel über Apoptose wurde erläutert, dass unterschiedliche extra- und intrazelluläre Signale eine Zelle in den programmierten Zellselbstmord treiben können. Das p53 zeigt hinsichtlich seiner Aktivierung ein vergleichbares Spektrum; p53 ist sogar in der Lage auf unterschiedliche Signale verschiedenartige Prozesse zu initiieren. Hier ein Überblick:
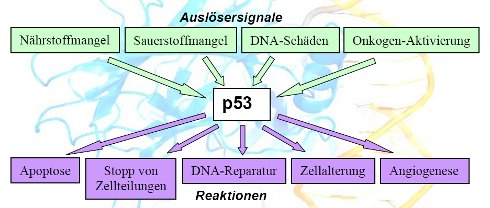
Wie kann ein einzelnes Protein so etwas leisten?
Unter Menschen würde man beispielsweise miteinander reden oder eine Nachricht schreiben. p53 kann zwar einiges, aber weder hören noch lesen. Dennoch werden intrazellulär z.B. von der Zelloberfläche in den Zellkern oder zwischen benachbarten Zellen Informationen versandt. Dies geschieht auf molekularer Ebene immer nach dem gleichen Prinzip: durch eine chemische Reaktion mit einem Enzym oder Zusammenlagerung verschiedener Moleküle wird die dreidimensionale Struktur eines Proteins verändert. Die Variationsbreite solcher so genannter posttranslationaler Modifikationen ist enorm: z.B.
- Phosphorylierung-Dephosphorylierung (Anfügen oder Entfernen einer Phosphat-Gruppe)
- Glykosylierung-Deglykosylierung (Anfügen oder Entfernen versch. Zuckerreste)
- Methylierung-Demethylierung (Anfügen oder Entfernen einer CH3-Gruppe)
- ... u.a.m.
Das p53 ist ein Paradebeispiel, wie unterschiedliche chemische Veränderungen an verschiedenen Stellen im Molekül unterschiedliche Prozesse in Gang setzen können. Beispiel einer Signalkaskade infolge von DNA-Schädigung:
- p53 ist in normalen Zellen aufgrund der Bindung an das Protein MDM2 inaktiv; p53 kommt nur in geringen Mengen vor, weil MDM2 eine chemische Veränderung bewirkt, wodurch p53 aus dem Zellkern transportiert wird und im Cytoplasma abgebaut wird.
- DNA-Schäden infolge von Strahlen oder Chemikalien aktivieren bestimmte Enzyme (= Kinasen wie ATM), die an p53 Phosphat-Gruppen binden (= phosphorylieren) -> MDM2 kann nicht länger an p53 binden -> p53-Moleküle bilden im Kern stabile Vierer-Einheiten (= Tetramere).
- p53-Tetramere sind Transkriptionsfaktoren: sie binden an bestimmte Stellen der DNA und stimulieren die Bildung weiterer Proteine (z.B. p21) -> p21 bindet an bestimmte Enzyme, die den Eintritt in die Zellteilung bewirken (z.B. CDK2), und inaktiviert sie.
- die Inaktivierung von CDK2 verhindert die Phosphorylierung des Proteins RB1 -> Zelle kann die G1-Phase des Zellzyklus nicht verlassen (Stopp jeglicher Zellteilungen).
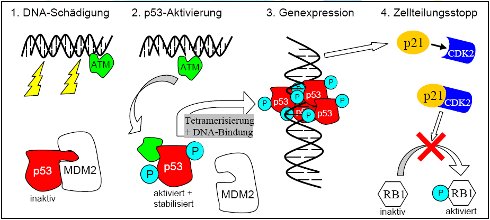
Solange Kinasen wie ATM infolge von DNA-Schäden aktiviert sind, wird der Zellteilungsstopp aufrechterhalten. Konnten DNA-Reparaturenzyme die Schäden beheben, wird diese Signalkaskade gestoppt und die Zelle kann sich wieder teilen. Für die Auslösung von Prozessen wie Apoptose gibt es vergleichbare Signalkaskaden, die jedoch durch andere Proteine und Enzyme gesteuert werden.
Glossar:
Dalton (Da) : nach dem englischen Naturforscher John Dalton benannte Masseeinheit, die bei Masseangaben von Biomolekülen verwendet wird (1Da = 1u = 1,660 x 10^27 kg; 1 Kilodalton = 1.000 u)
Gelelektrophorese : Methode zur Auftrennung von Gemischen elektrisch geladener hochmolekularer Stoffe (vor allem Proteine, Nukleinsäuren)
Domäne : Bereich eines Proteins mit definierter Funktion und Konformation innerhalb der Tertiärstruktur
Quellen (Dez. 2008):
http://en.wikipedia.org/wiki/p53
p53 in health and disease, K. H. Vousden and D. P. Lane, Nature Reviews, 2007, Vol. 8, S. 275 ff
