Teil 1 – CRISPR-Cas9 in der gentechnischen Anwendung
Sachanalyse
1. CRISPR-Cas als prokaryotisches Virenabwehrsystem
Nach einer Infektion einer Bakterienzelle durch Bakteriophagen wird virale DNA zunächst durch spezielle Enzyme der Bakterienzelle (Cas-Endonukleasen) geschnitten. Die entstehenden Fragmente viraler DNA werden dann in einen speziellen Bereich des bakteriellen Chromosoms, den CRISPR-Locus (clusterly regularly interspaced short palindromic repeats; Abbildung 1) ) eingebaut. Die in den Locus eingebauten viralen DNA-Fragmente werden als Protospacer bezeichnet. Sie sind jeweils durch kurze DNA-Abschnitte (Repeats) voneinander getrennt. Aus Protospacern und Repeats werden durch Transkription konstitutiv RNA-Moleküle (CRISPR-RNA; crRNA) gebildet (Abbildung 1).
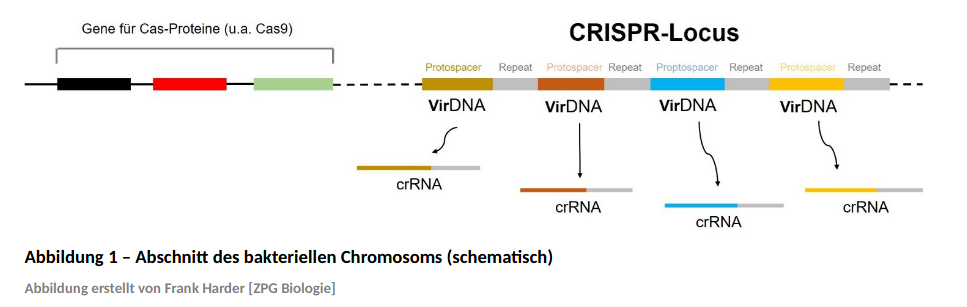
Abbildung erstellt von Frank Harder [ZPG Biologie]
Dies crRNA-Moleküle dienen der Aktivierung einer weiteren Cas-Endonuclease (Cas9-Endonuklease). Jedoch wird im natürlichen Virenabwehrprozess zur Aktivierung von Cas9 neben der crRNA noch ein weiteres Molekül benötigt. Diese Tracr-RNA wird aus einem von CRISPR unabhängigen Locus ebenfalls konstitutiv transkribiert und bindet dann am Repeat-Bereich der crRNA. Die entstehende RNA-Haarnadelstruktur bindet am Enzym Cas9 und führt zu dessen Aktivierung (Abbildung 2).
Wird eine Bakterienzelle durch eine zweite Infektion mit einem Bakteriophagen erneut mit dessen DNA konfrontiert, bindet der aktive CRISPR-Cas9-Komplex über den Protospacerbereich spezifisch an der bakteriellen DNA. In der Folge wird die virale DNA in diesem Bereich geschnitten. Dadurch kann das Virus sich in der Zelle nicht vermehren.
Bei CRISPR-Cas9 handelt es sich also um ein prokaryotisches Virenabwehrsystem, bei welchem Cas-Endonucleasen gezielt diejenige virale DNA schneiden, der die Zelle schon einmal ausgesetzt war.
Um zu verhindern, dass auch eigene DNA-Sequenzen geschnitten werden, muss Cas9 virale DNA sicher erkennen. Dies erfolgt über die Erkennung einer kurzen, charakteristischen DNA-Sequenz viraler DNA. Diese Sequenz wird als PAM (protospacer adjacent motif) bezeichnet. Erst in der Folge der Bindung eines Cas9-Moleküls an einer PAM-Sequenz wird an der Bindungsstelle die Doppelstrangstruktur aufgelöst und ein crRNA-tracrRNA-Molekül kann sich anlagern. Das Enzym Cas9 durchtrennt dann beide Stränge der Fremd-DNA an derselben Stelle (Abbildung 2). Cas9 braucht also sowohl die spezifische Erkennungssequenz als auch ein PAM auf der Ziel-DNA um binden und erfolgreich schneiden zu können. Da das Erbgut der Bakterien selektionsbedingt keine PAMs für seine Cas9-Proteine besitzt, ist es vor der Zerstörung durch das eigene Abwehrsystem geschützt.
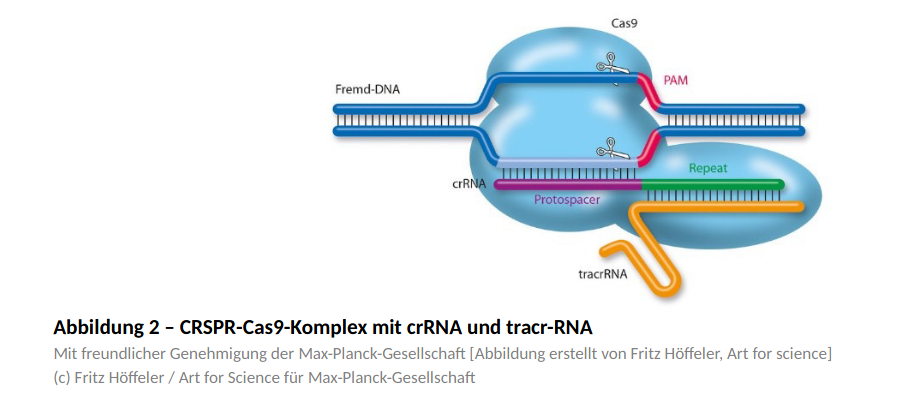
(c) Fritz Höffeler / Art for Science für Max-Planck-Gesellschaft
2.CRISPR-Cas9 in der gentechnischen Anwendung
Cas-Proteine aus verschiedenen Bakterienarten benötigen meist auch verschiedene PAM-Sequenzen. In der gentechnischen Anwendung sind daher heute auch verschiedene Cas9-Proteine mit unterschiedlichen PAM- Sequenzen verfügbar, um die Bandbreite möglicher Schnittsequenzen zu erhöhen. Um zielgerichtet DNA schneiden zu können, wird meist Cas9-mRNA zusammen mit guide-RNA per Mikroinjektion in die Zelle eingebracht. Guide- RNA besteht schon von vornherein aus allen Bestandteilen, die zur sequenzspezifischen Aktivierung von Cas9 benötigt werden (Protospacer-, Repeat- und tracr-RNA). Cas9-mRNA wird in der Zelle translatiert. Das Enzym bindet die guide-RNA und ist dann in der Lage, über die Sequenz der guide-RNA DNA spezifisch zu schneiden (Abbildung 3).
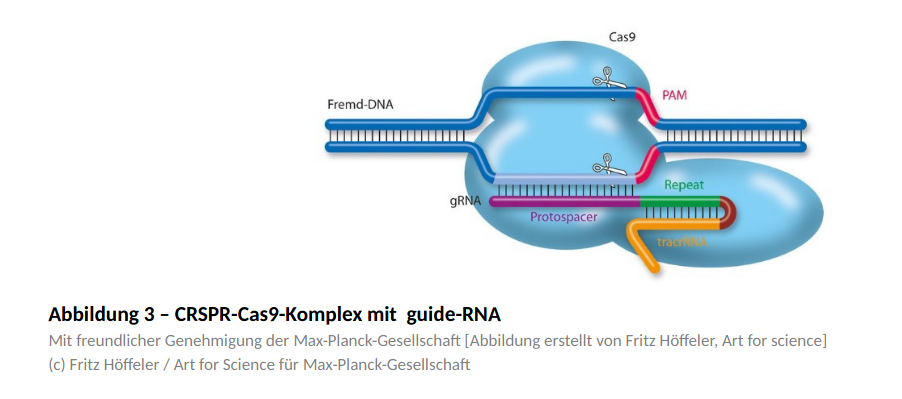
(c) Fritz Höffeler / Art for Science für Max-Planck-Gesellschaft
3. Reparatur von Doppelstrangschnitten
Doppelstrangschnitte der DNA können in Zellen auf natürliche Weise repariert werden (Abbildung 4). Zunächst werden enzymatisch kurze Einzelstrangbereiche hergestellt. Unter Verwendung komplementärer (homologer) Sequenzen des entsprechenden Bereichs des Schwesterchromosoms wird der Bereich dann weitläufig ergänzt. Der Prozess ähnelt in gewisser Weise den Abläufen der Replikation und wird als homologe Rekombination bezeichnet. Die homologe Rekombination ist jedoch nicht immer erfolgreich.
Die Reparatur des Doppelstrangbruchs kann aber auch zufällig unter Verlust von Nucleotiden erfolgen. Dies kann zum Knockout eines Gens (Gene knockout) und damit zum Fehlen oder Funktionsverlust des Genprodukts führen.
In Abhängigkeit vom Ziel des gentechnischen Prozesses sollen Gene ausgeschaltet oder gezielt mutiert werden. Bei der gezielten Mutagenese wird in großen Mengen Fremd-DNA zugegeben, deren Flanken zum Bereich der Schnittstelle homolog (komplementär) sind, die aber im Schnittbereich von der Originalsequenz abweicht. Diese DNA kann dann mit einer erhöhten Wahrscheinlichkeit anstelle des Schwesterchromosoms als Vorlage für die Reparatur durch homologe Rekombination verwendet werden und zum Einbau einer gewünschten Mutation in die DNA führen (Gene knockin).
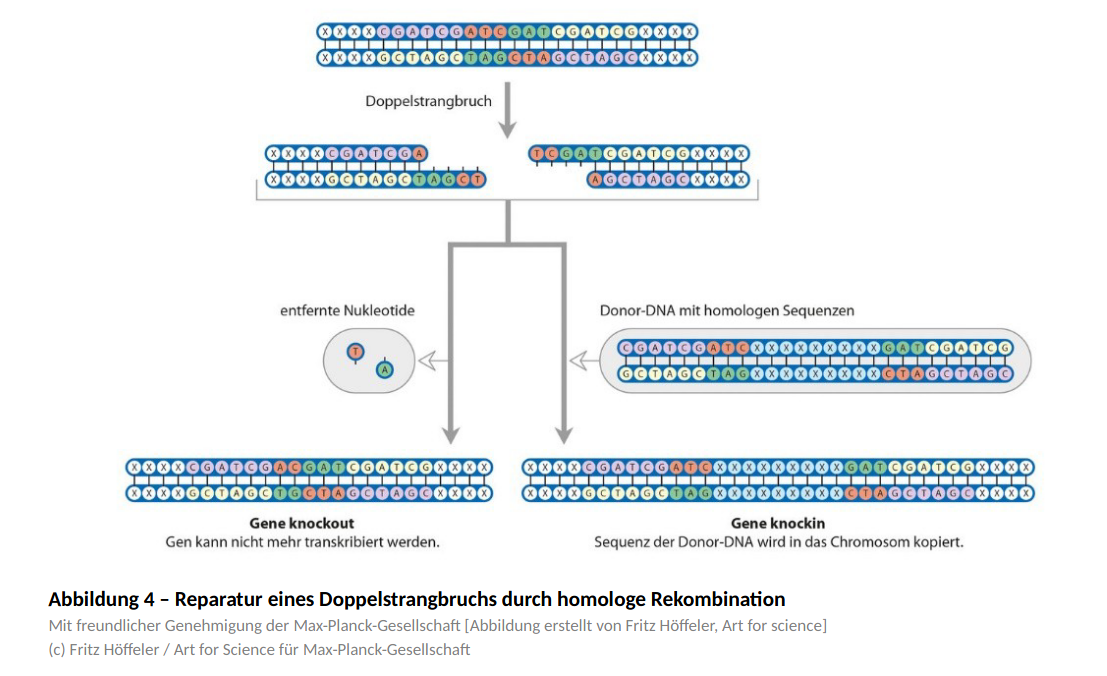
(c) Fritz Höffeler / Art for Science für Max-Planck-Gesellschaft
4. CRISPR-Cas9-Genedrive
Um die Verbreitung einer Mutation in einer Population über die statistische Verteilung gemäß der Mendelschen Regeln hinaus zu erhöhen, bedient man sich eines molekularbiologischen Tricks. Durch CRISPR-Cas9 und homologe Rekombination wird in die DNA einer Zelle zunächst ein DNA-Abschnitt (CRISPR-Cas9-Genedrive- Kassette) eingebaut, welcher unter Kontrolle eines Promotors sowohl das Cas9-Gen als auch einen Bereich für die notwendige guide-RNA enthält. Gelingt der Einbau dieses Bereichs durch homologe Rekombination auf einem der beiden Chromosomen, wird in der Folge Cas9 exprimiert und schneidet mit Hilfe der guide-RNA auch das Schwesterchromosom an der entsprechenden Stelle. Durch natürliche homologe Rekombination wird der Doppelstrangbruch im Schwesterchromosom mit Hilfe des schon mutierten Chromosoms repariert. Dadurch können mit einer gewissen Wahrscheinlichkeit homozygot mutierte Organismen entstehen, die ihre Allele sicher in die Folgegeneration weitergeben, wo sich der Prozess wiederholt. Der durch diesen Mechanismus erzielte Effekt auf das Vererbungsmuster wird ebenfalls als Genedrive bezeichnet.
Didaktische Reduktionen
Die im Material verwendeten Materialien weisen an einigen Stellen grundlegende Vereinfachungen und didaktische Reduktionen auf:
1. Differenzierung zwischen cr-RNA/tracr-RNA und guide-RNA
Ziel des Materials ist die Vermittlung der Hintergründe zur gentechnischen Anwendung von CRISPR-Cas9, nicht aber der Prozesse der bakteriellen Virenabwehr.
Daher wird auf die Verwendung der Begriffe crRNA und tracr-RNA verzichtet und stattdessen grundsätzlich der Begriff guide-RNA verwendet, auch in Material 3, bei dem es um die grundlegende Funktionsweise des Enzyms geht und eigentlich die Begriffe cr-RNA/tracr-RNA fachlich korrekt wären.
2. PAM-Sequenz
Die Notwendigkeit einer PAM-Sequenz führt in der gentechnischen Praxis zu gewissen Einschränkungen in der Auswahl möglicher Schnittstellen. Zum grundlegenden Verständnis der Arbeitsweise von CRISPR-Cas9 in der Gentechnik ist die Betrachtung einer PAM aber nicht zwingend vonnöten. Daher wurde in den Materialien die PAM- Sequenz an dieser Stelle nicht thematisiert. Cas9 bindet daher in den Materialien vereinfachend schon mit guide- RNA aktiviert an der DNA.
3. Homologe Rekombination
Die molekularen Abläufe während der homologen Rekombination sind sehr komplex und würden bei detaillierterer Betrachtung das Material enorm verkomplizieren, ohne dabei einen Mehrgewinn zu erzielen. Die homologe Rekombination wurde daher sehr stark vereinfacht dargestellt.
4. Erfolgsquote der genetischen Veränderung durch CRISPR-Cas9 und homologe Rekombination
Im Material wird vereinfachend davon ausgegangen, dass die genetische Veränderung der Anopheleseier durch CRISPR-Cas9 und homologe Rekombination im Labor, ebenso wie der Genedrive in Mücken, zuverlässig erfolgreich verläuft. Dies ist natürlich bei weitem nicht der Fall, würde aber die Verständlichkeit des Materials erheblich erschweren.
5. Experimentelle Untersuchung der Wirkung mutierter Mücken auf die Populationsentwicklung
Der suggerierten hohen Zuverlässigkeit des Genedrives ist auch die Abweichung des Materials bei der Untersuchung des Einflusses genetisch veränderter Anophelesmücken auf die Population geschuldet. Im Gegensatz zur in der Originalliteratur beschriebenen Untersuchung des Effekts, den heterozygot mutierte Männchen auf eine Versuchspopulation von Anophelesmücken haben, wird im Material von der Ausbringung homozygot mutierter männlicher Mücken ausgegangen. Da schon vorab von einer sehr hohen Zuverlässigkeit des Genedrive ausgegangen wurde, ist die Existenz heterozygoter Männchen nur unter Betrachtung statistischer Erfolgsquoten des Verfahrens möglich. Dies könnte in einer Nachbesprechung thematisiert werden.
Didaktische Anmerkungen
Flexibel einzusetzendes Material
Die Materialien zur Wirkungsweise von CRISPR-Cas9 (Material 3) und der grundlegenden Vorgehensweise beim GenomeEditing mittels CRISPR-Cas9 und homologer Rekombination (Material 4) sind so konzipiert, dass sie auch unabhängig von der Thematik der Unterrichtseinheit (Bekämpfung von Malaria) an anderer Stelle eingesetzt werden können, beispielsweise bei der genetischen Veränderung des Gens für den CCR5-Rezeptor in Knochenmarksstammzellen im Rahmen einer Gentherapie gegen HIV-Infektionen.
Binnendifferenzierung
Für die Materialien 2 und 5 liegen gestufte Hilfen vor.
Material 5 liegt zudem in zwei Niveaustufen (A und B) vor.
Unterrichtsgang: Herunterladen [docx][559 KB]
Unterrichtsgang: Herunterladen [pdf][574 KB]
Weiter zu Unterrichtsgang
